同変グラフ畳み込み拡散モデル(EDM: E(3) Equivariant Diffusion Model)による分子生成をtf2で実装します。
拡散モデルによる分子デザイン
拡散モデル(Diffusion Model)を利用した画像生成が、GPTなど大規模言語モデル(LLM)と並んで近年の生成AI(Generative AI)ブームを牽引しています。
ゆえに拡散モデル=画像生成というイメージを持たれがちですが、実際には拡散モデルは画像に限らず動画、音声、点群などあらゆる連続値データの生成において強力な手法です。とくに分子デザインは拡散モデルの有望な応用先の一つとして盛んに研究されています。
拡散モデルの優位性
2010年代にはVAEやGANに代表される深層学習ベースの分子自動生成手法が多く提案されましたが、どれも(例えばAlphafold2のような)汎用化学研究ツールとして広く普及するレベルには到達しませんでした*1。ここには大きく2つの課題があったと考えています。ひとつは人間のデザイン力が強い領域である低分子化合物までしかまともに生成できなかったこと、もうひとつは実務的な条件付け生成が容易でなかった*2ことです。
しかし、2020年代から急速に研究が進んだ拡散モデルでは、
① 学習安定性が高く、大きく複雑な構造生成が可能
② 高精度な条件付け生成により、実務的な分子デザインが可能
という優れた特性を持つために、これまでは”興味深い技術”どまりだった自動分子デザイン技術(私見)が、ついに実務研究ツールとして普及しつつあるように思います。
① 学習安定性が高く、大きく複雑な構造生成が可能
拡散モデルの学習はVAEやGANなどと比べて圧倒的に安定しています。ここには2つの理由があり、ひとつはVAEやGANでは2つのネットワークを協調的に訓練する必要がある一方で、拡散モデルではシンプルなロス関数で一つのネットワークを訓練すればよいだけだからということ。もう一つは拡散モデルの徐々にノイズ低減していくという生成アーキテクチャは難しい問題をより単純な部分問題の集合に自動分割する効果がある*3ことです。後者について、VAEやGANなどでは一発書きで高品質なお絵描きに挑戦していたのに対して拡散モデルではラフ画->線入れ->ベタ塗り->仕上げ塗りと工程を分けているようなものと喩えることができます。どちらが難しいかは明らかですね。

学習が安定だと何がうれしいかというと、大量のデータで長時間訓練できます。拡散モデルの表現力は非常に高いため大量のデータで長時間訓練すると複雑で巨大な構造も生成可能となります。分子デザインでいえば、これまでの生成モデル(VAEやGANなど)では低分子くらいまでしかまともに生成できなかったのが、拡散モデルであればより複雑で巨大な構造である中分子やタンパク質を高品質に生成することができます。これはうれしい。
② 高精度な条件付け生成により、実務的な分子デザインが可能
高品質な分子構造生成が可能だとしても、ランダムに構造が生成されだけならばそれはただの分子構造サイコロでしかありません。 実研究における分子デザインには常に制約が伴います。たとえば親水性を高めたい、基本骨格を指定したい、結合サイトにフィットするような形状にしたい…、このような状況で制御できない分子構造サイコロは役に立ちません。実用の鍵は条件付け生成です。
拡散モデルが条件付け生成においても優れた性能を発揮することは、Dall-EやStable Diffusionのようなテキスト条件付け拡散モデルがAIの非専門家ですら知られるようになったことからも明らかです。
なぜ拡散モデルでは高精度な条件付けが可能なのでしょうか?まず、拡散モデルの逆拡散プロセスでは画像にデノイジングネットワークを適用してノイズ低減することをT回繰り返して最終画像を生成します。ゆえに条件付けのチャンスもT回存在するために、与えた条件を正確に反映することができるようになるというのが大きな理由のひとつです*4。
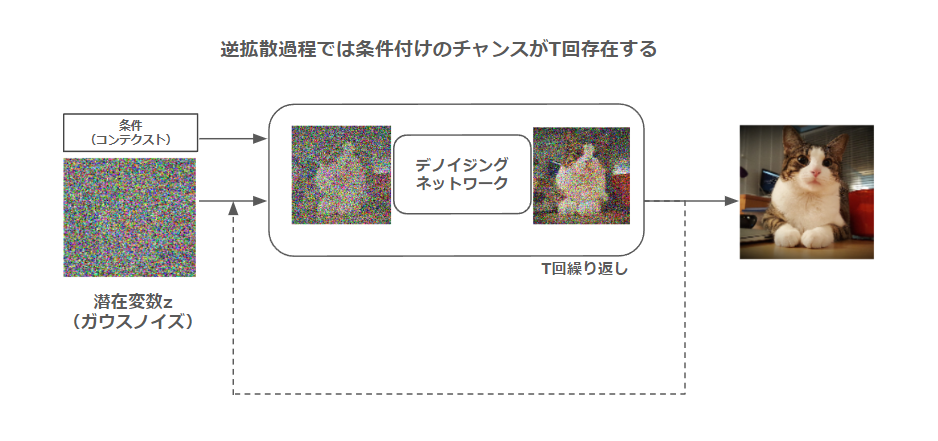
条件付けについて、画像でいえば生成画像を猫にしたいか/犬にしたいなどクラスラベルを条件として与えることができるほかに、画像の一部だけを与えて残りの部分を生成させるというような構造的な条件付け生成も可能です。
とくに化学ドメインにおいては親水性や求電子性のような物理化学的な特性による条件付けはもちろん、構造的な条件付け生成が可能になることが大きな意味を持ちます。たとえば、タンパク質結合サイトや触媒活性部位の構造にフィットする分子構造を直接デザインすることができるようになるなど様々なユースケースが考えられます。
創薬・材料科学分野で広がる応用
拡散モデルによる分子デザインのイントロの締めとして、個人的にimpressiveだった拡散モデル×化学ドメインの3つの研究を紹介します。
① RFDiffusionによる複合体デザイン
De novo design of protein structure and function with RFdiffusion | Nature
大きくて複雑な構造(タンパク質)を生成可能 & 高精度な条件付け(結合サイト立体構造による条件付け)という拡散モデルの強みが遺憾なく発揮されている研究です。これは詳細を語るよりもデモを見たほうがインパクトが分かりやすいでしょう。

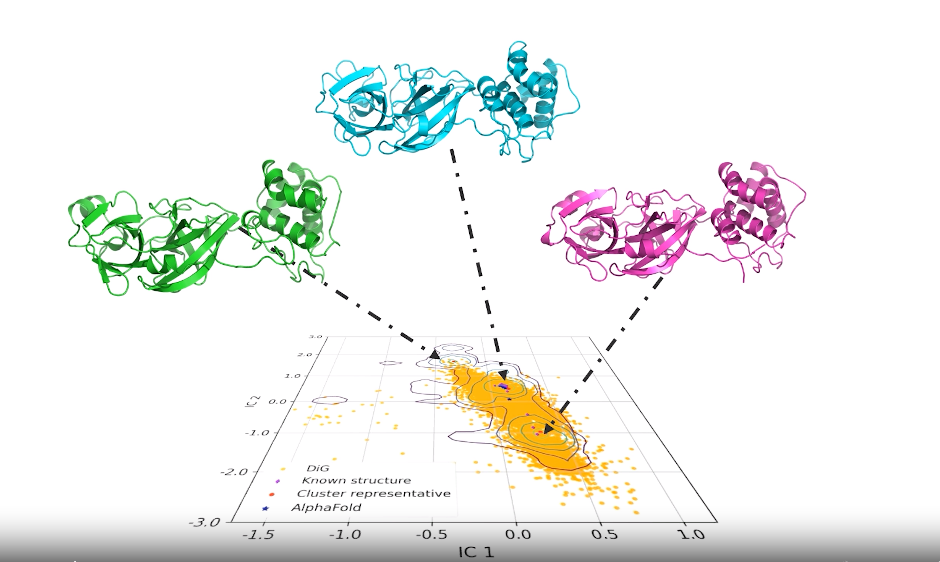
② Distributional Graphormerによる構造分布予測
[2306.05445] Towards Predicting Equilibrium Distributions for Molecular Systems with Deep Learning
Alphafold2などのように最安定立体構造を予測するのではなく、拡散モデルで立体構造分布を予測するMicrosoftの研究。タンパク質立体構造分布、タンパク質とリガンドの相互作用、触媒表面への分子吸着の3例で実証。分子統計力学を知らないとインパクトがわかりにくいのだけども、構造分布がわかればタンパク質とリガンドの結合の強さ=薬剤としての効力の強さが計算できるし、触媒表面におけるターゲット分子の構造分布がわかれば触媒の強さが計算できるので汎用性次第では化学企業のR&Dプロセスのゲームチェンジャーとなりうる。
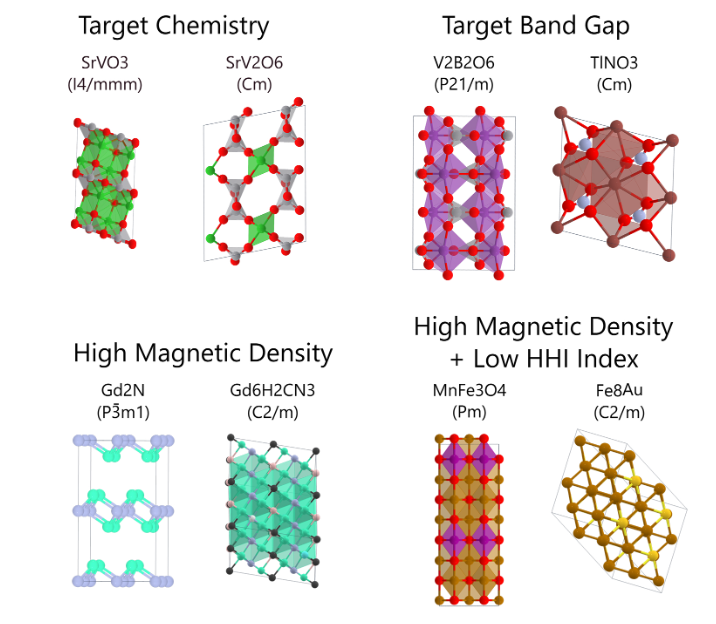
③MatterGen: 所望の特性をもつ結晶構造を生成
MatterGen: a generative model for inorganic materials design - Microsoft Research
従来の無機結晶材料開発の計算科学的アプローチといえば、安定な結晶構造をランダム探索 -> シミュレーションで物性を確認というガチャを無限に回してるみたいなイメージがあった(超私見)。それはあまりに非効率なので、Dall-Eに「ハムスターの画像を生成して」と指示するように、「伝導性の高い結晶構造生成して」と指示できる拡散モデルベースの結晶構造生成AI作りましたよ、というのがこの研究。明日にでも使えそうなほど実務的であるという観点でimpressiveだった。ちなみにこちらもMicrosoft。
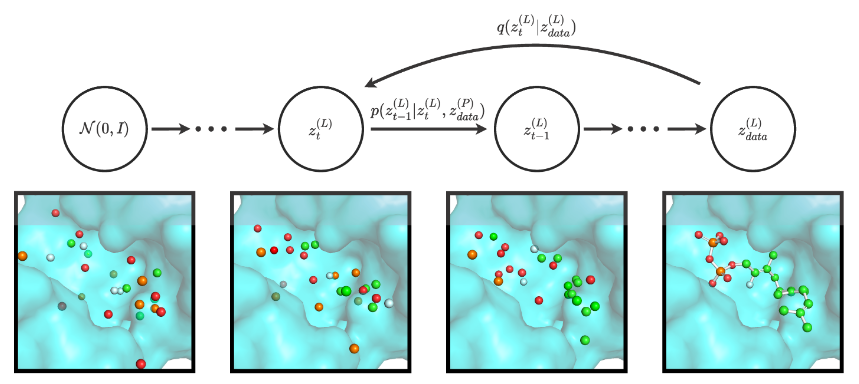
EDM:同変グラフ畳み込み拡散分子生成モデル
これまで見てきた例の通り、ここ数年の分子生成モデルは発展が速すぎてわけわからん状態になっていたので、本稿ではこの分野の源流的な手法の一つであるEDM( E(3) Equivariant Diffusion Model)を実装していきます。
この手法では並進・回転同変グラフ畳み込みネットワークと拡散モデルを組み合わせたことで高品質な立体構造生成を実現したことがポイントです。
同変グラフ畳み込みネットワーク
分子構造を表現する方法にはSMILESのようなテキスト表記、二次元グラフ、画像、距離行列などいろいろありますが、もっとも情報が失われないという観点であれば全原子3次元グラフで表現するのが最適でしょう。(分子はスティック&ボールではない?知らない話ですね...)
そうではあるのですが、単純な3Dグラフニューラルネットワークは入力構造の位置や向きのずれにめっぽう弱いという弱点があります。入力分子構造の向きを少し変えただけで生成結果が変わってしまうので不安定すぎて使い物になりません。いちおう、いわゆるデータ拡張(data augmentation)的なことをすればある程度は防げるのですがシンプルに非効率です。
ここで重要になるのが並進・回転同変性を備えたグラフニューラルネットワークです。
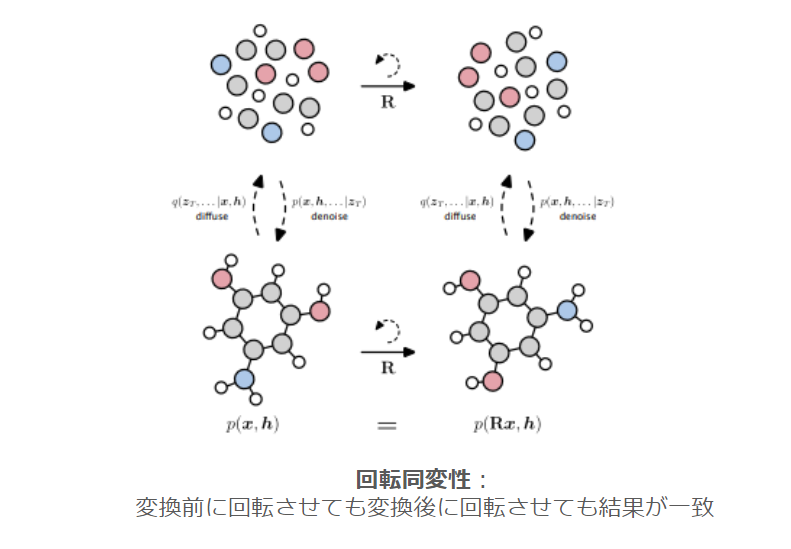
回転同変性を備えたグラフニューラルネットワークでは変換前の構造を回転させても変換後の構造を回転させても同じ結果が得られるため、無理筋なデータ拡張が不要であり学習効率が非常に高く、分子構造のような対称性を持つ3Dグラフ構造の学習に適しています。雑な喩えをするならば、並進回転同変性のないGCNNで分子構造を学習することは、CNNを使わず全結合層だけで画像を学習するくらい困難です。
[参考] 同変性について理解を深めたい方向け:
対称性は学習にどのように活かせられるか | 日経Robotics(日経ロボティクス)
ディープラーニングを支える技術〈2〉
Recruit Data Blog | NeurIPS 2021 参加報告 前編
並進・回転同変性の獲得
上述した通り、並進(位置のずれ)・回転(向きのずれ)に対する同変性獲得が3次元グラフニューラルネットワーク成功の鍵です。
①並進同変性の獲得
並進同変性の獲得については実はとても簡単であり、グラフニューラルネットワークの外で実現することができます。すなわちネットワークへの入力前に分子構造の重心を原点に合わせる変換を行うことで並進同変性は達成されます。そりゃそうじゃ。
②回転同変性の獲得
ちょっとややこしいのは回転同変性(向きのずれ)の獲得です。端的には回転変換の影響を受ける原子絶対座標は使わずに、回転変換の影響を受けない原子間の相対位置情報だけをネットワークに入力することによって回転同変性を獲得します。直感的にはPyMolで分子構造をぐるぐる回すと各原子の絶対位置は変わるけど原子間の相対的な位置関係は変わらないことからも、相対位置情報が回転変換に対して同変性を持つことをイメージすることができます。
具体的な更新式を以下に示します。

原子座標xと原子特徴hでそれぞれ別に更新を行っているのがポイントです。原子特徴hの更新について、原子間距離dやエッジ特徴aなどそもそも座標の取り方の影響を受けない値だけをネットワークに入力するので回転不変な更新になっています。原子座標xの更新について、右辺第一項は更新前座標ですが無変換なので回転同変です。右辺第二項は原子間ベクトル(x_i - x_j) を回転不変な値だけで算出されるスカラ値 で伸縮するだけの操作なので回転同変です。よって原子座標xの更新は回転同変な操作となります。
拡散モデルとの組み合わせ
拡散モデルとの組み合わせについて、拡散過程は回転不変なので逆拡散過程におけるノイズ予測に同変グラフニューラルネットワークを使うだけでOKです。拡散過程(ノイジングプロセス)が回転不変であることはガウスノイズの等方向性を考えれば納得できます。詳細は 拡散モデル データ生成技術の数理 4. 5 対称性を考慮した拡散モデル を参照ください。
TF2での実装
同変グラフ拡散モデルをTF2で実装します。
実装全文:
github.com
オフィシャル実装:
GitHub - ehoogeboom/e3_diffusion_for_molecules
QM9データセットの入手
今回は学習にQM9データセットを使います。QM9は最大 9 個の重原子 (C, O, N, F) で構成される約 134,000 個の分子立体構造データセットであり、各分子の立体構造はDFTで最適化されています。なお、QM9は存在しうる分子構造の列挙であり合成可能性や安定性は一切考慮されていないため、ファンタジーな構造が多く含まれることに留意が必要です。
入手先はいろいろあるのですが今回は DeepChemが配布しているsdf形式データセット をダウンロードして使用します。
参考:
future-chem.com
分子構造をネットワーク入力用にフォーマット
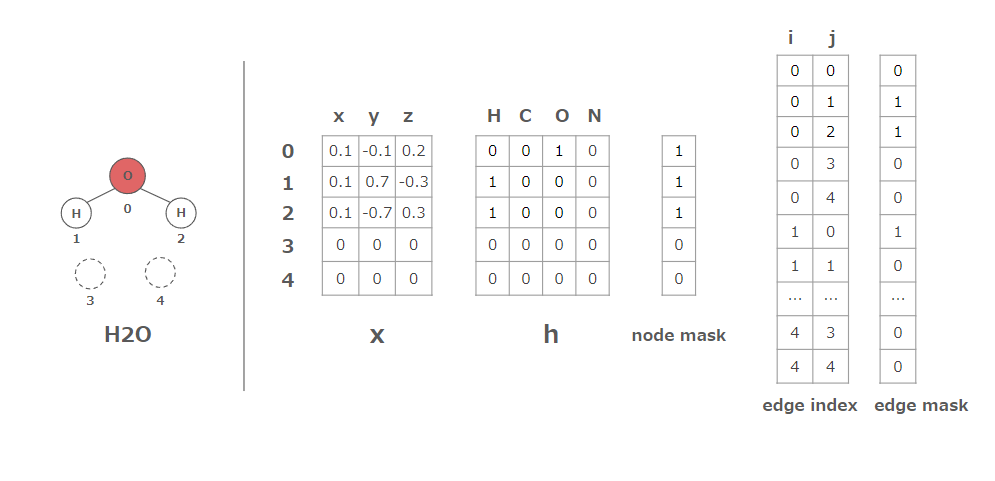
QM9の分子構造をネットワーク入力用にフォーマットしtfrecord形式で保存します。ひとつの分子構造から以下に示す5つの行列が作成されます。
- x: 原子のxyz座標
- h: 原子タイプのonehot表現
- node_mask: ダミー原子用のマスク
- edge_index: すべての原子対ijの組み合わせを列挙したインデックス
- edge_mask: 自己エッジ(i==j)およびダミー原子を含むエッジのマスク
ダミー原子はNLPでいうpaddingであり、ミニバッチ内で原子数を揃えるために導入します。
同変グラフ畳み込みネットワーク
論文に書いてある通り実装するだけです。
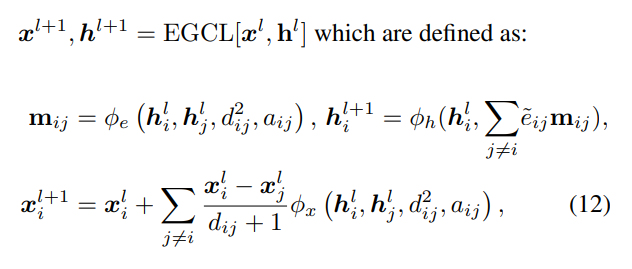
ノードごとに総和をとる処理(segment_sum_by_node)についてだけはコードがやや煩雑ですが、やりたいことはgroupby(indices_i).sum()というだけなので難しいことをしているわけではありません。
拡散モデルのトレーニング
ノイズ予測ネットワークには同変GCNNを使いますが、拡散モデル自体はごく普通にDDPMを実装すればOKです。逆拡散プロセスについても同様。
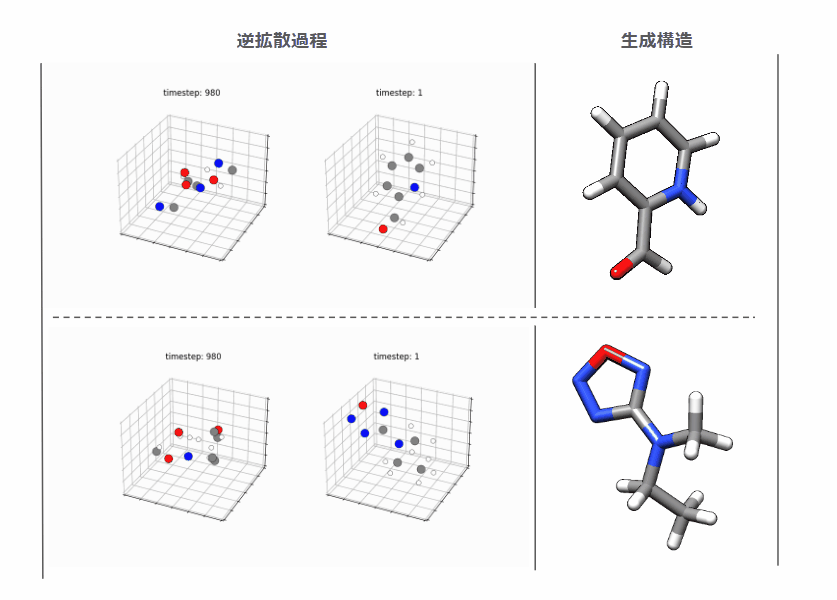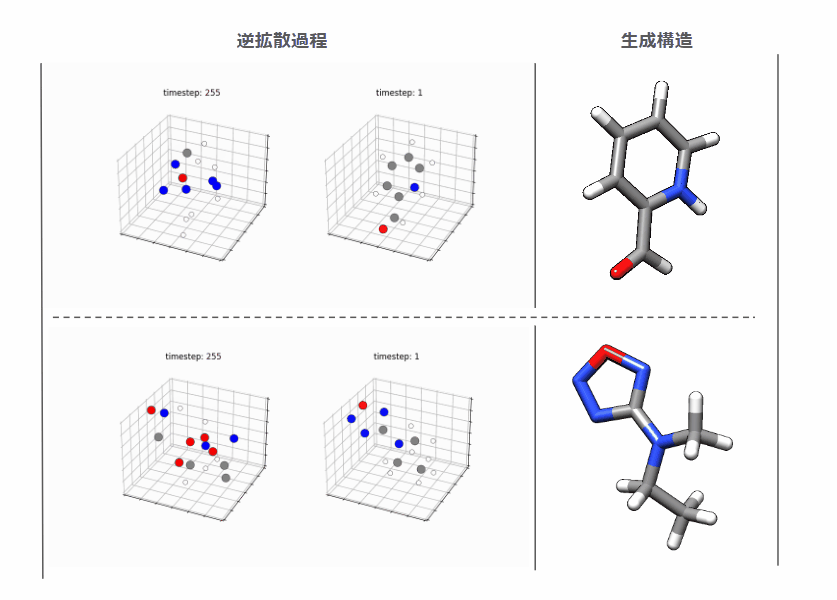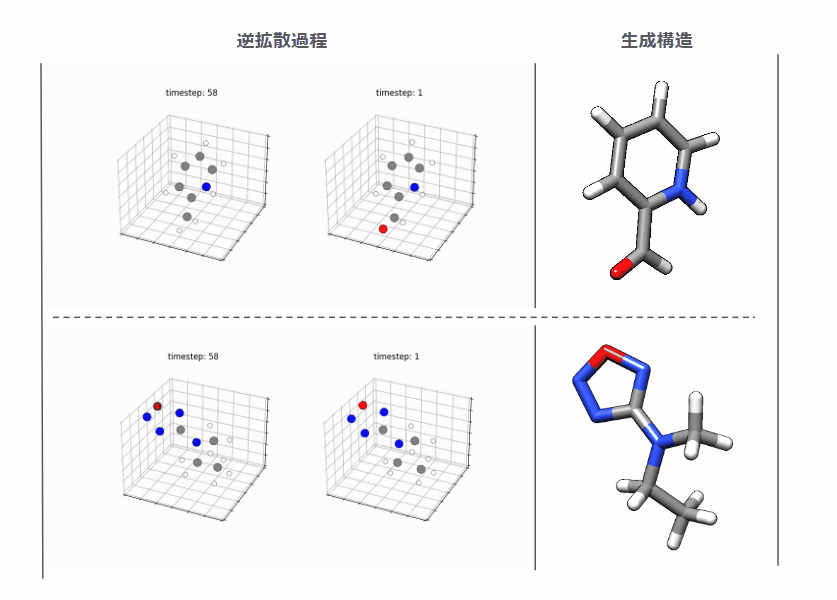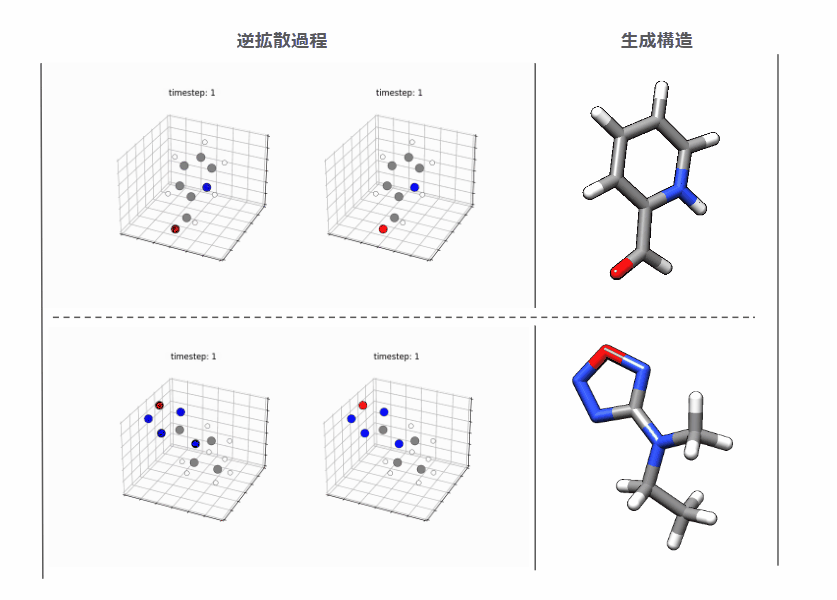
生成結果

結合数/結合距離に破綻のない3次元構造が直接生成されており、拡散モデルの生成品質の高さに驚きました。
とはいえ、やたら環をまいた化合物などファンタジーな構造もよく出力するので上の例ではチェリーピックして自然に見える化合物を選んでます。*5これはそもそもQM9データセットが合成可能性を考慮していないファンタジー寄りなデータセットなのでモデルというよりは学習したデータ側に理由があると考えています。実用性重視なら商用化合物カタログとかにデータセットを変えたほうがよいのでしょう。
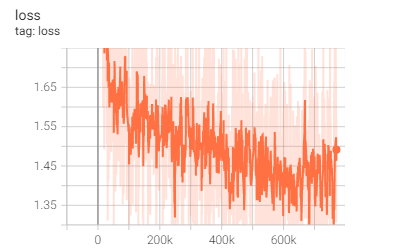
ちなみに学習時間はRTX4080で24時間くらいです。ロスの減少からも安定した学習ができていることがわかります。
参考文献
コンピュータビジョン最前線 Summer 2023
わずか30Pの解説にDDPMのエッセンスが凝縮されていて大変理解しやすく、実装時のリファレンスとして最適。
コンピュータビジョン最前線 Winter 2023
夏号では説明があまりなかった条件付け生成や高速化手法について補足が行われている。セットでどうぞ。
拡散モデル データ生成技術の数理
PFN岡之原氏による本格派の拡散モデル解説書。統計力学に深く関連するデノイジングスコアマッチングから拡散モデルの説明を始めるので分子シミュレーション屋さんには理解しやすいと思われる。ギブスエナジー。
ディープラーニングを支える技術〈2〉
5.3節に不変性、同変性の説明あり。