DeepChem②(sklearnモデルで回帰)
DeepChemのチュートリアルを参考に、化合物の水溶解度データセット(delaneyデータセット)を用いて単目的回帰します。
化合物フィンガープリントによる単目的回帰
まずはオーソドックスな化合物フィンガープリントによって単目的回帰を行ってみます。 内部的にはrdkitのMorganFingerprintを呼び出しているようです。
データ読み込みと分割
import deepchem as dc ### データセット読み込み csv = "datasets/delaney-processed.csv" featurizer = dc.feat.CircularFingerprint(size=1024) loader = dc.data.CSVLoader( tasks=['measured log solubility in mols per litre'], smiles_field="smiles", featurizer=featurizer) dataset = loader.featurize(csv) ### データ分割 splitter = dc.splits.ScaffoldSplitter(dataset) train_dataset, valid_dataset, test_dataset = splitter.train_valid_test_split(dataset,frac_train=0.8, frac_valid=0.1,frac_test=0.1)
前回同様の手順でcsvデータを読み込み、化合物構造に基づいてデータを分割しました。 実問題では化合物構造が偏りがちなのでこのような処理が簡単にできるのはうれしいですね。
一応、rdkitを用いてデータ取り出しのテストを行います。
### 描画テスト from rdkit import Chem from rdkit.Chem.Draw import IPythonConsole mols = [] for smile,lable in zip(train_dataset.ids[:8],train_dataset.y[:8]): print(lable,smile) mol = Chem.MolFromSmiles(smile) mols.append(mol) img=Draw.MolsToGridImage(mols,molsPerRow=4,subImgSize=(200,200)) img
SVR回帰
前処理として目的変数の標準化をします。
### 目的変数のみ標準化 ### train_datasetを標準化の基準に指定 transformers = [dc.trans.NormalizationTransformer(transform_y=True, dataset=train_dataset)] for transformer in transformers: train_dataset = transformer.transform(train_dataset) valid_dataset = transformer.transform(valid_dataset) test_dataset = transformer.transform(test_dataset)
valid_datasetを用いてハイパーパラメータサーチを行います。 テストなので探索範囲は適当です。
import numpy as np from sklearn.svm import SVR def skmodel_builder(model_params, model_dir): sklearn_model = SVR(**model_params) return dc.models.SklearnModel(sklearn_model, model_dir) params_dict = {"kernel": ["linear","rbf"], "C": np.linspace(0.1,10,10)} metric = dc.metrics.Metric(dc.metrics.r2_score) optimizer = dc.hyper.HyperparamOpt(skmodel_builder,verbose=False) best_model, best_model_hyperparams, all_model_results = optimizer.hyperparam_search(params_dict, train_dataset, valid_dataset, transformers, metric=metric)
精度検証
test_datasetで予測精度の検証をします。
from deepchem.utils.evaluate import Evaluator test_evaluator = Evaluator(best_model, test_dataset, transformers) test_r2score = test_evaluator.compute_model_performance([metric])
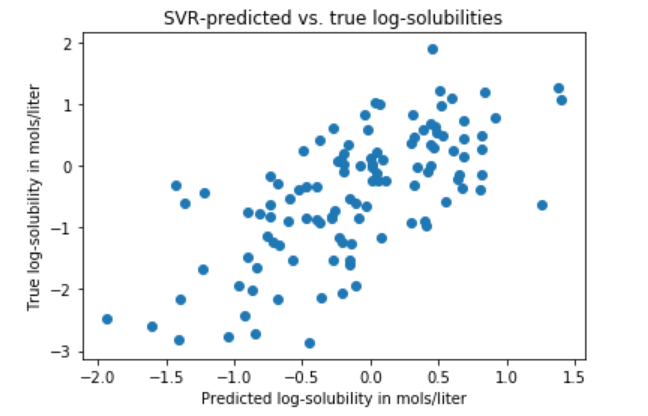
### out computed_metrics: [0.34182224998573496]
なんとも言えない精度ですが、可視化までしてみましょう。
import matplotlib.pyplot as plt %matplotlib inline sk = "measured log solubility in mols per litre" predicted_test = best_model.predict(test_dataset) true_test = test_dataset.y plt.scatter(predicted_test, true_test) plt.xlabel('Predicted log-solubility in mols/liter') plt.ylabel('True log-solubility in mols/liter') plt.title('SVR-predicted vs. true log-solubilities')